A/B test
- 두 개의 그룹을 비교하여 차이가 있는지 판단
- A/B test, T-test… 등으로 불림
임의화(randomnization)
공정해야 한다라는 것의 의미는?- 실험에서 고려한 의도적인 차이에 의한 효과(처리효과,treatment 효과)외에
- 다른 요인의 차이에 의해 두 그룹간에 효과의 차이가 나타날 가능성이 있으면 안된다는 것.
- ex) 신약의 효과를 입증하고자 하는 실험. 신약과 기존약의 차이외에 다른 요인이 없어야함.(인종,성별,나이,…)
- 두 그룹간의 비교가 공정하지 못하다면?
- 효과가 차이가 날 경우 진짜 의도한 차이에 의한 효과가 아니기에 과대평가 할 수 있음.
- 효과가 차이가 안날 수도 있는데 이 경우 과소평가.
- 두 경우 모두, 실험에서 고려한 의도적인 차이가 결과에 미치는 영향을 정확히 파악할 수 없음.
- 목적 : 두 그룹에 대한 비교는 반드시
공정해야 함. 이때 공정함을 구현하는 방법. - 어떻게 구현?
- 두 개의 처리 중 하나를 실험대상에게 지정(배정)할때
- 처리의 배정을 임의로 결정(randomly assigned treatment) 하자.
- 평균적으로 처리 외의 다른 요인들이 두 그룹에서 유사하게 나타나게 할 수 있음.
- 두 그룹이 유사하기 때문에 처리외의 다른 요인으로 인한 효과의 차이가 나타날 가능성이 적음.
임의화 실험의 예시
임의화 구현
- 1874명의 사람들을 임상실험에 참가하는 환자들이라 가정하고 임의화 실험을 해보자
url = "https://ilovedata.github.io/teaching/bigdata2/data/physical_test_2018_data.csv"
physical_data = pd.read_csv(url)
twogroup = physical_data[["TEST_AGE","TEST_SEX","ITEM_F001","ITEM_F002"]].rename(columns = {"ITEM_F001":"height","ITEM_F002":"weight"})
twogroup| TEST_AGE | TEST_SEX | height | weight | |
|---|---|---|---|---|
| 0 | 33 | M | 159.2 | 57.2 |
| 1 | 48 | F | 155.8 | 52.9 |
| 2 | 22 | M | 175.2 | 96.2 |
| 3 | 29 | M | 178.7 | 79.4 |
| 4 | 31 | F | 160.1 | 50.2 |
| ... | ... | ... | ... | ... |
| 1869 | 18 | M | 179.8 | 79.8 |
| 1870 | 29 | F | 156.3 | 51.4 |
| 1871 | 19 | M | 173.9 | 64.5 |
| 1872 | 66 | F | 141.3 | 54.9 |
| 1873 | 51 | F | 155.5 | 59.9 |
1874 rows × 4 columns
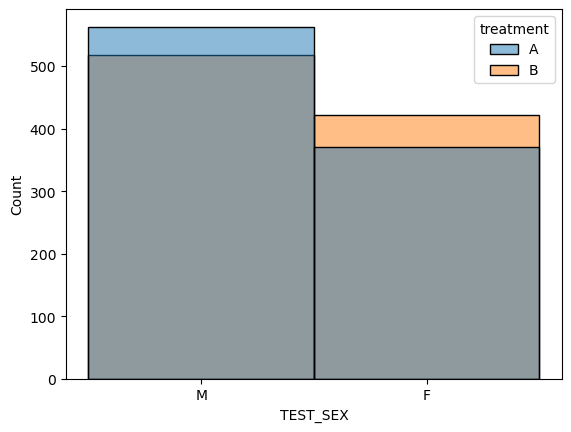
- 각 환자에게 A,B 중 어떤 약을 투약할지 임의로 지정!
array(['A', 'A', 'A', ..., 'A', 'A', 'A'], dtype='<U1')| TEST_AGE | TEST_SEX | height | weight | treatment | |
|---|---|---|---|---|---|
| 0 | 33 | M | 159.2 | 57.2 | A |
| 1 | 48 | F | 155.8 | 52.9 | A |
| 2 | 22 | M | 175.2 | 96.2 | B |
| 3 | 29 | M | 178.7 | 79.4 | A |
| 4 | 31 | F | 160.1 | 50.2 | A |
| 5 | 23 | F | 157.8 | 60.1 | A |
| 6 | 11 | M | 165.5 | 60.3 | B |
| 7 | 24 | M | 174.9 | 74.5 | A |
| 8 | 18 | M | 181.0 | 71.3 | B |
| 9 | 41 | F | 160.6 | 72.7 | B |
- 임의화로 지정한 두 그룹에 대하여 나이,몸무게,키에 대한 분포 비교
| TEST_AGE | height | weight | |||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| count | mean | std | min | 25% | 50% | 75% | max | count | mean | ... | 75% | max | count | mean | std | min | 25% | 50% | 75% | max | |
| treatment | |||||||||||||||||||||
| A | 934.0 | 35.616702 | 17.432484 | 11.0 | 21.0 | 29.0 | 50.75 | 84.0 | 934.0 | 167.455353 | ... | 174.5 | 195.2 | 934.0 | 67.466381 | 13.359358 | 34.1 | 57.925 | 66.85 | 75.300 | 132.3 |
| B | 940.0 | 35.329787 | 16.892562 | 11.0 | 22.0 | 28.5 | 48.00 | 83.0 | 940.0 | 166.716064 | ... | 173.0 | 190.6 | 940.0 | 66.154362 | 12.926716 | 31.1 | 56.400 | 64.65 | 75.025 | 122.6 |
2 rows × 24 columns
<AxesSubplot:xlabel='TEST_AGE', ylabel='Count'>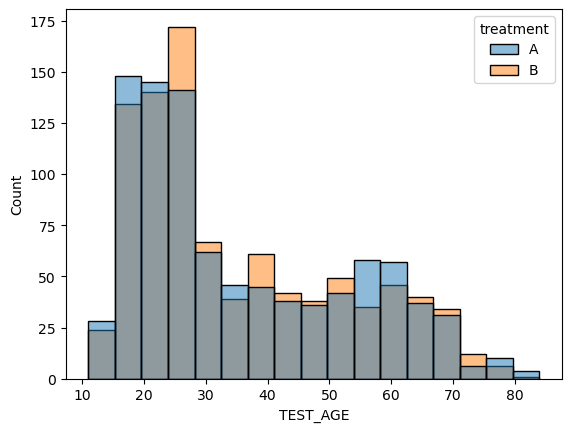
<AxesSubplot:xlabel='weight', ylabel='Count'>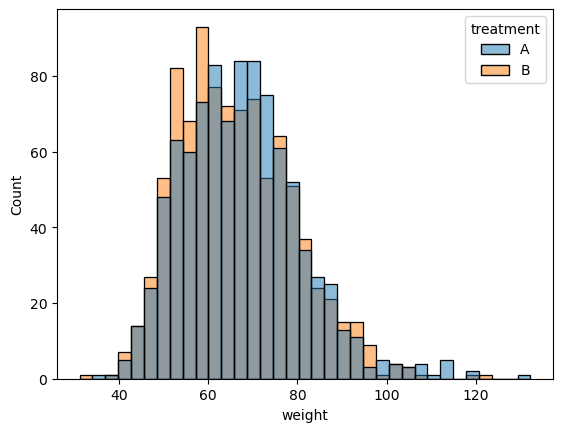
<AxesSubplot:xlabel='TEST_AGE', ylabel='Count'>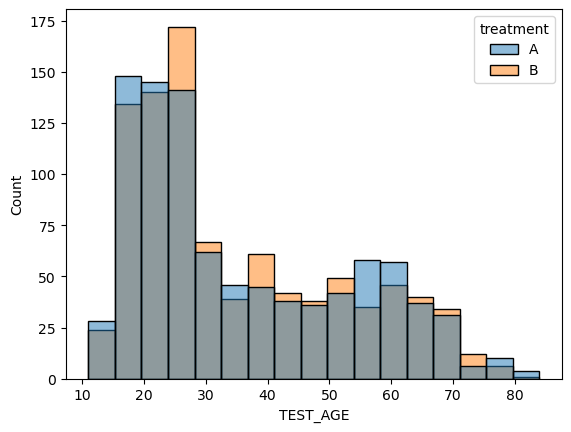
결과
- 처리를 제외한 다른 요인들의 분포가 각각의 그룹에서 유사하게 나타냄
- 처리를 제외한 다른 요인들로 인한 효과의 차이가 나타날 가능성 거의 없음.
- “실험에서 처리를
임의화를 통하여 배정하면 두 그룹을공정하게 비교할 수 있음”을 확인!
A/B test
- 두 그룹간의 차이가 있는지 비교.
- 유의성 검정을 통해 수행할 수 있음.
filename = "https://ilovedata.github.io/teaching/bigdata2/data/drug.csv"
three_drug_wide = pd.read_csv(filename)
three_drug_wide.head(10)| Placebo | Old | New | |
|---|---|---|---|
| 0 | 31 | 23 | 23 |
| 1 | 28 | 17 | 17 |
| 2 | 34 | 29 | 11 |
| 3 | 36 | 23 | 11 |
| 4 | 33 | 17 | 9 |
| 5 | 27 | 17 | 16 |
| 6 | 39 | 22 | 16 |
| 7 | 25 | 17 | 14 |
| 8 | 23 | 23 | 17 |
| 9 | 29 | 25 | 18 |
| Placebo | Old | New | |
|---|---|---|---|
| count | 20.000000 | 20.000000 | 20.000000 |
| mean | 31.550000 | 20.900000 | 14.850000 |
| std | 4.334379 | 5.990343 | 4.319783 |
| min | 23.000000 | 5.000000 | 7.000000 |
| 25% | 28.750000 | 17.000000 | 11.000000 |
| 50% | 31.500000 | 21.000000 | 15.000000 |
| 75% | 34.500000 | 24.250000 | 17.000000 |
| max | 39.000000 | 33.000000 | 24.000000 |
<class 'pandas.core.frame.DataFrame'>
RangeIndex: 20 entries, 0 to 19
Data columns (total 3 columns):
# Column Non-Null Count Dtype
--- ------ -------------- -----
0 Placebo 20 non-null int64
1 Old 20 non-null int64
2 New 20 non-null int64
dtypes: int64(3)
memory usage: 608.0 bytes- pd.melt(frame,id_vars,value_vars,var_name,value_name)
- frame : long형으로 바꿀 df
- value_vars : value가 될 variable. 즉 값으로 바꿀 열들
- var_name : “variable” column이 이름. variable컬럼에는 column이름들이 포함되어 있음
- value_name : “value” column의 이름. value column에는 값들이 들어 있음.
three_drug_long = pd.melt(frame = three_drug_wide,value_vars = ["Placebo","Old","New"],var_name = "treatment",value_name = "Value")
three_drug_long.sample(10)| treatment | Value | |
|---|---|---|
| 41 | New | 17 |
| 18 | Placebo | 30 |
| 46 | New | 16 |
| 17 | Placebo | 32 |
| 11 | Placebo | 36 |
| 55 | New | 17 |
| 48 | New | 17 |
| 51 | New | 7 |
| 0 | Placebo | 31 |
| 16 | Placebo | 30 |
cond = three_drug_long[three_drug_long.treatment == "Old"].index
two_drug = three_drug_long.drop(index=cond_col).reset_index(drop=True)
two_drug.head(10)| treatment | Value | |
|---|---|---|
| 0 | Placebo | 31 |
| 1 | Placebo | 28 |
| 2 | Placebo | 34 |
| 3 | Placebo | 36 |
| 4 | Placebo | 33 |
| 5 | Placebo | 27 |
| 6 | Placebo | 39 |
| 7 | Placebo | 25 |
| 8 | Placebo | 23 |
| 9 | Placebo | 29 |
| Value | ||||||||
|---|---|---|---|---|---|---|---|---|
| count | mean | std | min | 25% | 50% | 75% | max | |
| treatment | ||||||||
| New | 20.0 | 14.85 | 4.319783 | 7.0 | 11.00 | 15.0 | 17.0 | 24.0 |
| Placebo | 20.0 | 31.55 | 4.334379 | 23.0 | 28.75 | 31.5 | 34.5 | 39.0 |
| treatment | Value | |
|---|---|---|
| 0 | New | 14.85 |
| 1 | Placebo | 31.55 |
x_p = float(temp[temp.treatment == "Placebo"].Value)
x_n = float(temp[temp.treatment == "New"].Value)
test_stat = x_p - x_n
test_stat16.700000000000003def diff_mean(df,group_label,treatments = None):
"""
df를 받아서 검정통계량을 계산해주는 함수. 검정통계량은 두 그룹간의 평균의 차이
df : dataframe
group_label : 처리가 적용된 변수명(그룹화 레이블)
treatments : 비교하고 싶은 두 개의 처리명
return : A 그룹 평균 - B 그룹 평균
"""
df_group = df.groupby(group_label)
temp = df_group.mean().reset_index()
test_stat = float(temp[temp[group_label] == treatments[0]].Value) - float(temp[temp[group_label] == treatments[1]].Value)
return test_stat
diff_mean(three_drug_long,"treatment",treatments = ["Placebo","New"])16.700000000000003| treatment | Value | permuted_treatment | |
|---|---|---|---|
| 0 | Placebo | 31 | New |
| 1 | Placebo | 28 | New |
| 2 | Placebo | 34 | Placebo |
| 3 | Placebo | 36 | Placebo |
| 4 | Placebo | 33 | Placebo |
| 5 | Placebo | 27 | Placebo |
| 6 | Placebo | 39 | New |
| 7 | Placebo | 25 | New |
| 8 | Placebo | 23 | Placebo |
| 9 | Placebo | 29 | New |
| 10 | Placebo | 36 | Placebo |
| 11 | Placebo | 36 | Placebo |
| 12 | Placebo | 32 | New |
| 13 | Placebo | 30 | New |
| 14 | Placebo | 33 | Placebo |
| 15 | Placebo | 39 | Placebo |
| 16 | Placebo | 30 | New |
| 17 | Placebo | 32 | Placebo |
| 18 | Placebo | 30 | New |
| 19 | Placebo | 28 | New |
| 20 | New | 23 | Placebo |
| 21 | New | 17 | New |
| 22 | New | 11 | New |
| 23 | New | 11 | New |
| 24 | New | 9 | New |
| 25 | New | 16 | New |
| 26 | New | 16 | Placebo |
| 27 | New | 14 | Placebo |
| 28 | New | 17 | Placebo |
| 29 | New | 18 | Placebo |
| 30 | New | 10 | New |
| 31 | New | 7 | Placebo |
| 32 | New | 14 | New |
| 33 | New | 24 | New |
| 34 | New | 18 | Placebo |
| 35 | New | 17 | Placebo |
| 36 | New | 14 | New |
| 37 | New | 14 | Placebo |
| 38 | New | 16 | Placebo |
| 39 | New | 11 | New |
treatment = two_drug.treatment
two_drug_permuted = two_drug
random_permuted_treatment = treatment.sample(frac=1.0,replace=False).reset_index(drop=True)
#reset_index안하면 sample로 shuffle을 해도 안섞임! 아마 인덱스끼리 매칭해주는 기능 있는 듯.
random_permuted_treatment.head(10)0 New
1 New
2 Placebo
3 New
4 Placebo
5 New
6 Placebo
7 New
8 New
9 New
Name: treatment, dtype: objecttwo_drug_permuted = two_drug
two_drug_permuted["permuted_treatment"] = random_permuted_treatment
two_drug_permuted| treatment | Value | permuted_treatment | |
|---|---|---|---|
| 0 | Placebo | 31 | New |
| 1 | Placebo | 28 | New |
| 2 | Placebo | 34 | Placebo |
| 3 | Placebo | 36 | New |
| 4 | Placebo | 33 | Placebo |
| 5 | Placebo | 27 | New |
| 6 | Placebo | 39 | Placebo |
| 7 | Placebo | 25 | New |
| 8 | Placebo | 23 | New |
| 9 | Placebo | 29 | New |
| 10 | Placebo | 36 | Placebo |
| 11 | Placebo | 36 | New |
| 12 | Placebo | 32 | Placebo |
| 13 | Placebo | 30 | Placebo |
| 14 | Placebo | 33 | New |
| 15 | Placebo | 39 | Placebo |
| 16 | Placebo | 30 | Placebo |
| 17 | Placebo | 32 | New |
| 18 | Placebo | 30 | Placebo |
| 19 | Placebo | 28 | Placebo |
| 20 | New | 23 | Placebo |
| 21 | New | 17 | Placebo |
| 22 | New | 11 | New |
| 23 | New | 11 | New |
| 24 | New | 9 | New |
| 25 | New | 16 | New |
| 26 | New | 16 | New |
| 27 | New | 14 | Placebo |
| 28 | New | 17 | New |
| 29 | New | 18 | Placebo |
| 30 | New | 10 | Placebo |
| 31 | New | 7 | New |
| 32 | New | 14 | New |
| 33 | New | 24 | Placebo |
| 34 | New | 18 | Placebo |
| 35 | New | 17 | New |
| 36 | New | 14 | Placebo |
| 37 | New | 14 | New |
| 38 | New | 16 | Placebo |
| 39 | New | 11 | Placebo |
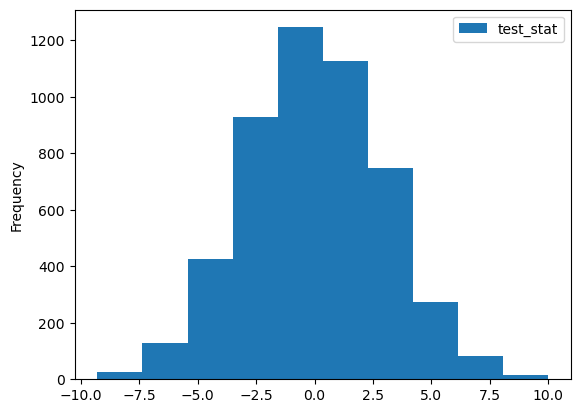
B = 5000
test_stat_permuted = pd.DataFrame({"test_stat":np.zeros(B)})
treatment = two_drug.treatment
for i in np.arange(B):
random_permuted_treatment = treatment.sample(frac=1.0,replace=False).reset_index(drop=True)
two_drug_permuted = two_drug
two_drug_permuted["permuted_treatment"] = random_permuted_treatment
test_stat_permuted.loc[i,"test_stat"] = diff_mean(two_drug_permuted,"permuted_treatment",["Placebo","New"])